Un modelo de aprendizaje profundo identifica una nueva y poderosa droga que puede matar muchas especies de bacterias resistentes a los antibióticos.
Usando un algoritmo de aprendizaje automático, los investigadores del MIT han identificado un nuevo y poderoso compuesto antibiótico. En pruebas de laboratorio, la droga mató a muchas de las bacterias más problemáticas del mundo que causan enfermedades, incluyendo algunas cepas que son resistentes a todos los antibióticos conocidos. También eliminó infecciones en dos modelos diferentes de ratón.
El modelo informático, que puede analizar más de cien millones de compuestos químicos en cuestión de días, está diseñado para detectar posibles antibióticos que matan bacterias utilizando mecanismos diferentes a los de los medicamentos existentes.
"Queríamos desarrollar una plataforma que nos permitiera aprovechar el poder de la inteligencia artificial para iniciar una nueva era de descubrimiento de fármacos antibióticos. Nuestro enfoque reveló esta asombrosa molécula que es posiblemente uno de los antibióticos más poderosos que se han descubierto",
dijo James Collins, profesor de Termeer de Ingeniería Médica y Ciencias en el Instituto de Ingeniería Médica y Ciencias (IMES) y el Departamento de Ingeniería Biológica del MIT.
En su nuevo estudio, los investigadores también identificaron varios otros candidatos prometedores a antibióticos, que planean probar más a fondo. Creen que el modelo también podría utilizarse para diseñar nuevos medicamentos, basándose en lo que han aprendido sobre las estructuras químicas que permiten que los medicamentos maten a las bacterias.
"El modelo de aprendizaje de la máquina puede explorar, en silico, grandes espacios químicos que pueden ser prohibitivamente caros para los enfoques experimentales tradicionales",
dijo Regina Barzilay, la Profesora de Electrónica Delta de Ingeniería Eléctrica y Ciencias de la Computación en el Laboratorio de Ciencias de la Computación e Inteligencia Artificial (CSAIL) del MIT.
Barzilay y Collins, que son codirectores de la facultad de la Clínica Abdul Latif Jameel de Aprendizaje Automático en Salud (J-Clinic) del MIT, son los autores principales del estudio, que aparece hoy en Cell. El primer autor del trabajo es Jonathan Stokes, un postdoctorado del MIT y del Instituto Broad del MIT y de Harvard.
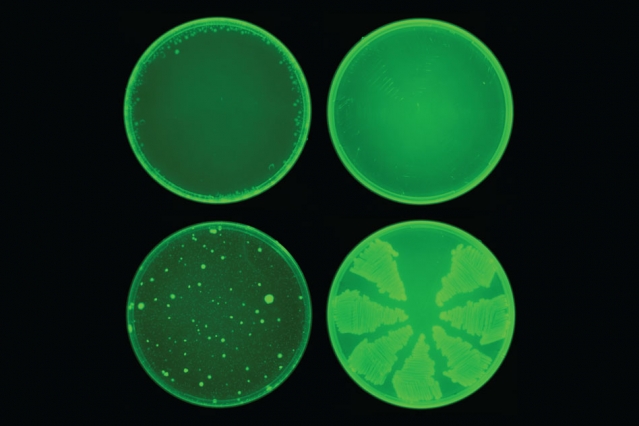
Los investigadores del MIT utilizaron un algoritmo de aprendizaje automático para identificar una droga llamada halicina que mata muchas cepas de bacterias. La halicina (fila superior) previno el desarrollo de la resistencia a los antibióticos en el E. coli, mientras que la ciprofloxacina (fila inferior) no lo hizo.
Imagen: cortesía del laboratorio Collins del MIT
En los últimos decenios, se han desarrollado muy pocos antibióticos nuevos, y la mayoría de esos antibióticos recientemente aprobados son variantes ligeramente diferentes de los medicamentos existentes. Los métodos actuales de detección de nuevos antibióticos suelen ser prohibitivos, requieren una importante inversión de tiempo y suelen limitarse a un estrecho espectro de diversidad química.
"Nos enfrentamos a una crisis creciente en torno a la resistencia a los antibióticos, y esta situación se está generando tanto por el creciente número de patógenos que se están volviendo resistentes a los antibióticos existentes, como por una reserva anémica en las industrias biotecnológica y farmacéutica para nuevos antibióticos",
dice Collins.
Para tratar de encontrar compuestos completamente novedosos, se asoció con Barzilay, el profesor Tommi Jaakkola y sus estudiantes Kevin Yang, Kyle Swanson y Wengong Jin, quienes han desarrollado previamente modelos informáticos de aprendizaje automático que pueden ser entrenados para analizar las estructuras moleculares de los compuestos y correlacionarlas con rasgos particulares, como la capacidad de matar bacterias.
La idea de utilizar modelos informáticos de predicción para la detección "in silico" no es nueva, pero hasta ahora, estos modelos no eran lo suficientemente precisos para transformar el descubrimiento de drogas. Anteriormente, las moléculas se representaban como vectores que reflejaban la presencia o ausencia de ciertos grupos químicos. Sin embargo, las nuevas redes neuronales pueden aprender estas representaciones automáticamente, mapeando las moléculas en vectores continuos que se utilizan posteriormente para predecir sus propiedades.
En este caso, los investigadores diseñaron su modelo para buscar las características químicas que hacen que las moléculas sean eficaces para matar a E. coli. Para ello, entrenaron el modelo en unas 2.500 moléculas, incluyendo unos 1.700 fármacos aprobados por la FDA y un conjunto de 800 productos naturales con diversas estructuras y una amplia gama de bioactividades.
Una vez que el modelo fue entrenado, los investigadores lo probaron en el Centro de Reposición de Drogas del Instituto Broad, una biblioteca de cerca de 6.000 compuestos. El modelo eligió una molécula que se predijo que tenía una fuerte actividad antibacteriana y que tenía una estructura química diferente a la de cualquier antibiótico existente. Utilizando un modelo diferente de aprendizaje por máquina, los investigadores también mostraron que esta molécula probablemente tendría una baja toxicidad para las células humanas.
Esta molécula, a la que los investigadores decidieron llamar halicina, por el sistema de inteligencia artificial ficticio de "2001: Una Odisea del Espacio", ha sido investigada previamente como posible droga para la diabetes. Los investigadores la probaron contra docenas de cepas bacterianas aisladas de pacientes y cultivadas en platos de laboratorio, y encontraron que era capaz de matar a muchas que son resistentes al tratamiento, incluyendo Clostridium difficile, Acinetobacter baumannii, y Mycobacterium tuberculosis. La droga funcionó contra todas las especies que probaron, con la excepción de Pseudomonas aeruginosa, un patógeno pulmonar difícil de tratar.
Para probar la eficacia de la halicina en animales vivos, los investigadores la usaron para tratar ratones infectados con A. baumannii, una bacteria que ha infectado a muchos soldados estadounidenses destacados en Irak y Afganistán. La cepa de A. baumannii que utilizaron es resistente a todos los antibióticos conocidos, pero la aplicación de un ungüento que contiene halicina eliminó completamente las infecciones en 24 horas.
Estudios preliminares sugieren que la halicina mata las bacterias al interrumpir su capacidad de mantener un gradiente electroquímico a través de sus membranas celulares. Este gradiente es necesario, entre otras funciones, para producir ATP (moléculas que las células utilizan para almacenar energía), de modo que si el gradiente se rompe, las células mueren. Este tipo de mecanismo de muerte podría ser difícil para que las bacterias desarrollen resistencia, dicen los investigadores.
"Cuando se trata de una molécula que probablemente se asocia con componentes de la membrana, una célula no puede necesariamente adquirir una sola mutación o un par de mutaciones para cambiar la química de la membrana externa. Mutaciones como esa tienden a ser mucho más complejas de adquirir evolutivamente",
dice Stokes.
En este estudio, los investigadores encontraron que la E. coli no desarrolló ninguna resistencia a la halicina durante un período de tratamiento de 30 días. Por el contrario, la bacteria comenzó a desarrollar resistencia al antibiótico ciprofloxacino en uno o tres días, y después de 30 días, la bacteria fue unas 200 veces más resistente a la ciprofloxacina que al principio del experimento.
Los investigadores planean realizar más estudios de la halicina, trabajando con una compañía farmacéutica u organización sin fines de lucro, con la esperanza de desarrollarla para su uso en humanos.
Después de identificar la halicina, los investigadores también utilizaron su modelo para examinar más de 100 millones de moléculas seleccionadas de la base de datos ZINC15, una colección en línea de unos 1.500 millones de compuestos químicos. Este análisis, que sólo tomó tres días, identificó 23 candidatos que eran estructuralmente diferentes de los antibióticos existentes y se predijo que no eran tóxicos para las células humanas.
En pruebas de laboratorio contra cinco especies de bacterias, los investigadores encontraron que ocho de las moléculas mostraban actividad antibacteriana, y dos eran particularmente poderosas. Los investigadores ahora planean probar estas moléculas más a fondo, y también revisar más la base de datos ZINC15.
Los investigadores también planean usar su modelo para diseñar nuevos antibióticos y optimizar las moléculas existentes. Por ejemplo, podrían entrenar el modelo para añadir características que harían que un antibiótico particular se dirija sólo a ciertas bacterias, evitando que mate a las bacterias beneficiosas del tracto digestivo de un paciente.
"Este trabajo innovador significa un cambio de paradigma en el descubrimiento de antibióticos y, de hecho, en el descubrimiento de fármacos en general",
dice Roy Kishony, profesor de biología e informática en Technion (el Instituto de Tecnología de Israel), que no participó en el estudio.
"Más allá de las pantallas de sílice, este enfoque permitirá utilizar el aprendizaje profundo en todas las etapas del desarrollo de los antibióticos, desde el descubrimiento hasta la mejora de la eficacia y la toxicidad a través de las modificaciones de los fármacos y la química medicinal".
La investigación fue financiada por la Clínica Abdul Latif Jameel de Aprendizaje Automático en Salud, la Agencia de Reducción de Amenazas de Defensa, el Instituto Broad, el Programa Make-It de DARPA, los Institutos Canadienses de Investigación en Salud, la Fundación Canadiense para la Innovación, el Programa de Cátedras de Investigación de Canadá, el Programa de Becas Banting, el Programa Científico de la Frontera Humana, la Fundación de la Plaza Pershing, la Fundación Nacional Suiza para la Ciencia, un premio para los primeros investigadores de los Institutos Nacionales de Salud, el Programa de Becas de Investigación para Graduados de la Fundación Nacional para la Ciencia y un regalo de Anita y Josh Bekenstein.